TogoDX/Humanの使い方
統合データ探索の原理
- DBCLSではこれまでに、様々な生命科学データを統一されたフォーマット(RDF形式)で集約したポータルサイト、RDF Portalの構築を行ってきました。これを利用することで、これまで個別に公開されていた遺伝子、タンパク質、立体構造、相互作用、化合物、糖鎖、疾患、バリアントを含む8つのカテゴリーに関わる20のデータベースから、65のAttributeを収集しました(2023年8月現在)。
- 収集した各Attributeは遺伝子IDや化合物IDといったそれぞれ個別のデータセットIDで整理されており、これらのデータの統合探索を実現するために、システム内では随時データセット間でID変換を行っています。
- ID変換に用いられるデータセット間のリンクは、生命科学のデータセット間のIDの関係を収録したID変換サービス、TogoIDのデータに基づいて構築されており、各データセットペアにおいて適切な変換経路を設定しています(図1)。
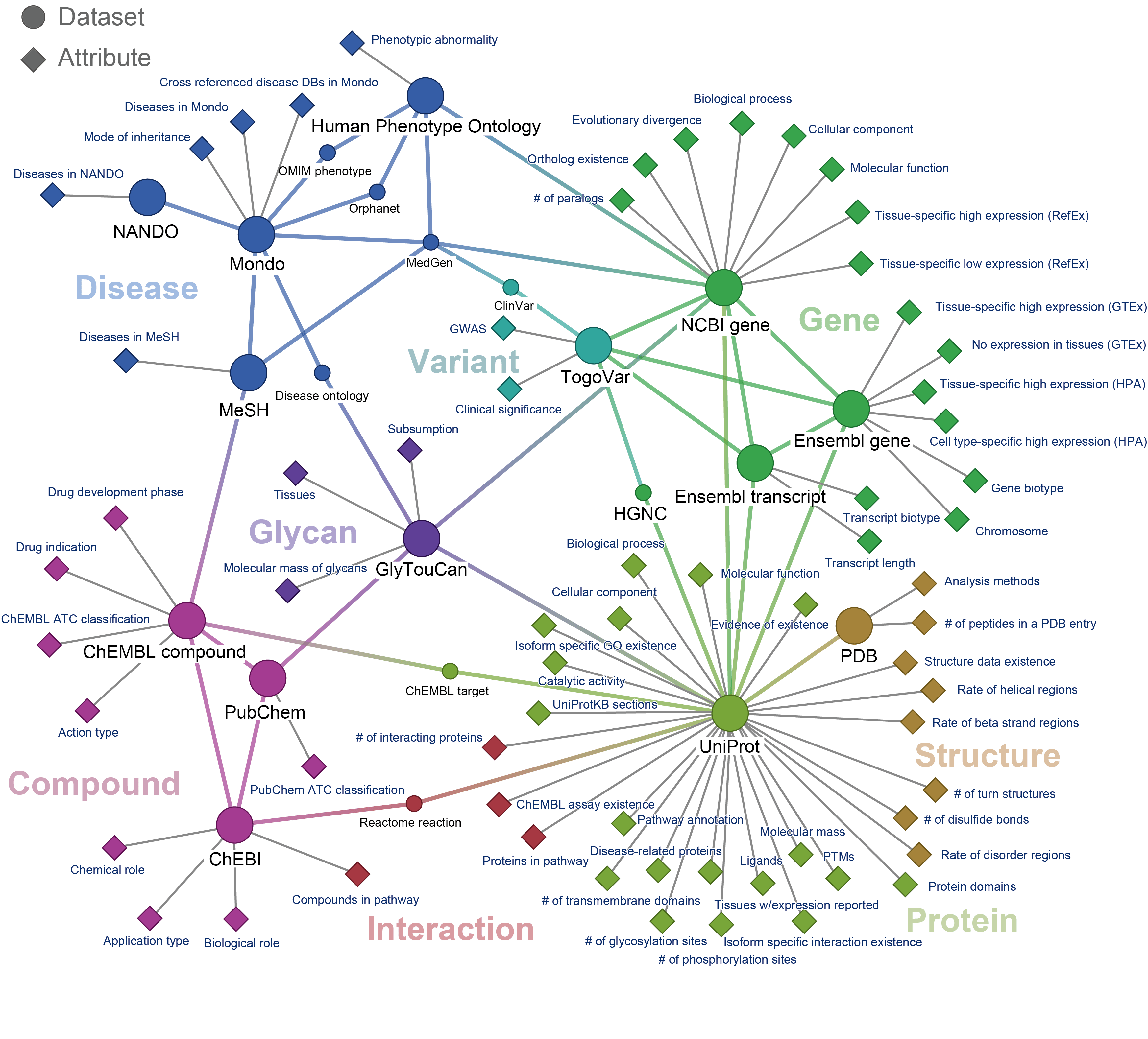
- 図1. TogoDX/Humanに含まれるデータセットおよびAttributeの関係図
- 大きな丸は対象となるデータセットを表しており、それらの間にID変換のためのリンクが張られている。 四角形はAttributeを示しており、繋がっているデータセットのIDをキーとしている。8種類の色は、データセットもしくはAttributeが属するカテゴリーを表している。 この図は、多様なAttributeがキーとなっているID間のリンクを通じて、統合されていることを示している。
- TogoDX/Humanで使用されているデータセット間のIDリンク関係の図
トップページの構成とAttributeの操作

- 図2. TogoDX/Humanのトップページから俯瞰できる各Attributeの分布や内訳
- Explorer画面(トップページ)では、遺伝子発現パターン、タンパク質の局在などの特徴あるアノテーションを
"Attribute"として表現しています(図2)。
- 初期状態ではあらかじめ選択された代表的なAttributeのみが表示されていますが、カテゴリー毎にAttributeを追加したり、Attributeのプリセットを選択することでユーザーに即したAttributeをカスタマイズして表示することが可能になっており、カスタマイズした情報はブラウザに記憶されます(図2A)。
- 各Attributeは、項目の構成(内訳)の100%積み上げグラフで表現されており、ユーザーは全データを俯瞰しながら興味あるAttributeを用いてデータを絞り込んだり、データセット全体のうちアノテーションされていない割合を把握することができます。
- Attributeは展開表示することが可能となっており、階層分類の場合はカラムブラウザが表示され、下位階層での分布を確認することができ(図2B)、連続値分布の場合はヒストグラム表示による直感的な分布の確認が可能です(図2C)。
絞り込みによるデータ探索とその結果の取得
- 対象とするデータセットを
"Select target dataset" から選択します(図2D)。
- 選択したデータセットを軸としたIDリンクを用いて、データ探索が行われます。
"Filtering" では、各Attributeの分類や値を選択することでデータの絞り込み条件を設定でき、条件を満たすデータセットのIDリストを取得することができます(図2E)。
- カラムブラウザからは下位階層を絞り込み条件に追加することができます(図2B)。
- ヒストグラムからは範囲を選択することで、絞り込み条件に追加することができます(図2C)。
"Projection" では、Filteringで絞り込まれたデータセットIDに対し、興味のあるAttribute階層をマッピングすることで、IDリストにおける分布を調べることができます(図2F)。
- これによって新たな発見やデータ解釈の可能性を高めることができます。
- FilteringやProjectionの操作を行うと、画面下方に条件を満たすデータセットIDのプレビューの表が現れ、条件を追加するごとに随時カラムが追加されます。
- 表上部の矢印アイコン、もしくは
"View results"をクリックすることでResults画面に切り替わります(図2G)。
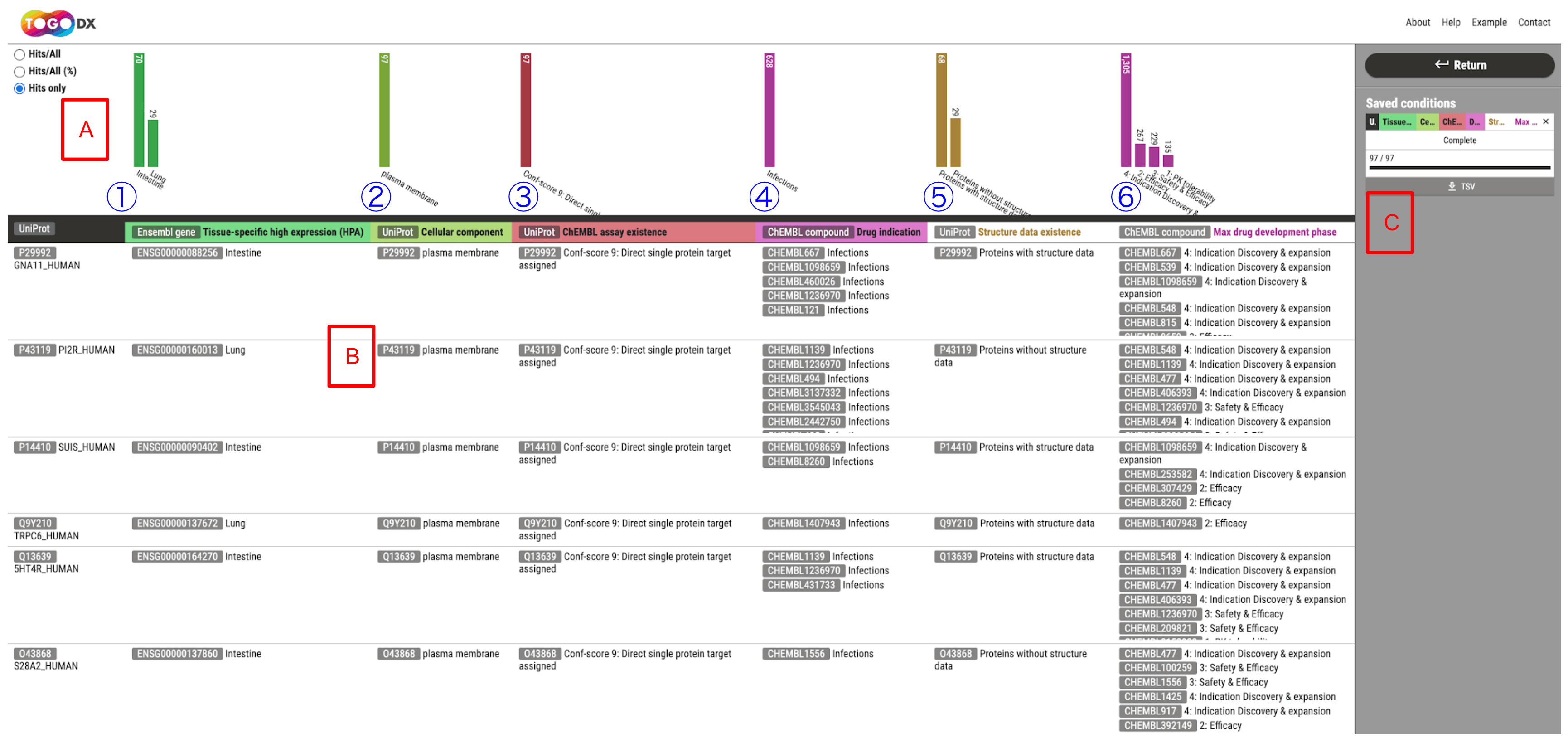
- 図3.
"Filtering"および"Projection"機能を用いた絞り込みによるデータ探索結果の例
"Results" 画面では、絞り込み条件に合致する対象データセットIDの一覧と、Filteringに用いたAttribute情報およびProjectionで指定したAttributeの内訳を表として閲覧できます(図3)。- 表の上部には、検索結果の内訳のグラフが表示され、視覚的に分布状況を知ることができます(図3A)。
- 各内訳の総数における絞り込み条件を満たすIDの数、その割合(%)、絞り込み条件を満たすIDの実数に表示を切り替えることで、分布や偏りを評価することができます。
- 各セルに表示されるデータセットIDをクリックすることで、そのIDに関連する詳細なアノテーション情報を確認できます(図3B)。
- 探索結果はTSVファイルとしてダウンロードすることができるので、JupyterLabやGoogle Colaboratory などの解析環境を利用し、さらに詳細なデータ解析を行うことができます(図3C)。
- TogoDX/Humanの検索条件はJSON形式ファイルとして入出力可能で、他のユーザーと共有して再現することができます。
〈この操作によるデータ探索の例〉
「類似した発現状況を示す遺伝子・タンパク質を標的とする医薬品の開発状況を調べる」
- 複雑な検索条件をひとつのアプリケーションで実現できるのがTogoDX/Humanの特長の一つです。
- この例では、以下の条件を満たすヒトのタンパク質の一覧を取得します。
- ① 肺または腸で組織特異的に高い遺伝子発現が確認
- ② タンパク質として細胞膜表面に局在
- ③ 何らかのタンパク質と化合物の直接の相互作用を検出する方法のデータが存在
- ④ 「感染」に対する薬効がある化合物に関わる
- ①〜④ の検索条件を各Attributeから探してfiltering条件として追加するだけで、すべての条件を満たす97個のタンパク質のIDリストが得られます(図3)。
- 続いて、Projection機能を使って、絞り込みで得られたタンパク質群が他のAttributeや階層の一部分においてどのような分布を示すかを調べることができます。
- この例では、以下の条件に基づく分類 を行っています。
- ⑤ PDBにおけるタンパク質立体構造データの有無
- ⑥ ChEMBLにおけるすべての適応症における化合物の最高の開発段階(研究段階、第1-3相、上市後)
IDマッピングによるデータ俯瞰
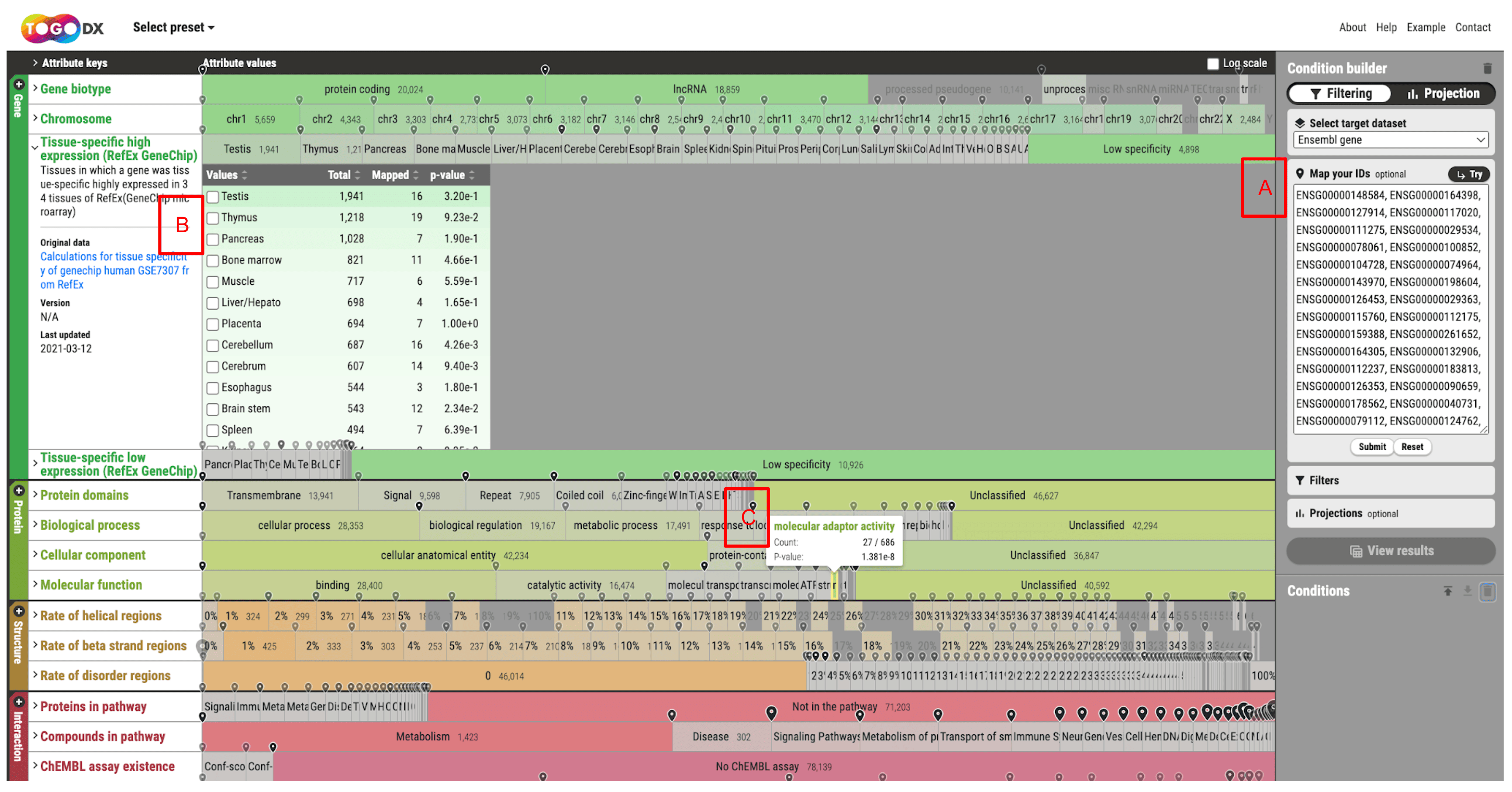
"Map your IDs" では、ユーザーの持つIDリストを入力することで各Attributeに対して簡易的なエンリッチメント解析を行うことが可能です。- IDリストをテキストエリアに入力しSubmitする(図4A)と、各AttributeごとにID変換が行われ、それぞれの内訳に対応するIDの数とp値が計算されます(図4B)。
- 対応するIDを含む内訳にはピンが表示され、また該当するIDが含まれない内訳はグレーアウトされ、分布や偏りを視覚的に確認することができます。また、絞り込みのためのFilterの一つとして利用することも可能です。
〈この操作によるデータ探索の例〉
COSMICに収載されているがんとの因果関係が示唆されている変異を含む遺伝子の分布や偏りを調べる
- COSMIC (Catalogue Of Somatic Mutations In Cancer) はがんと関連する体細胞変異の情報を集積したデータベースです。
- これらは、TogoDX/Human上における Ensembl gene ID のMap your IDsの実行例として
"Try" ボタンから利用可能です(図4A)。
- 例えば、この158遺伝子がどの組織で特異的に高発現しているかの分類をすぐに確認することができます(図4B)
- また、Gene Ontologyを使って分子機能別に分類することも容易です(図4C)。
その他
ページヘッダ
About
Help
- TogoDX/Humanの用語説明や機能説明へのリンク
Example
- TogoDX/Humanの具体的な事例解説へのリンク
Contact
初期化
- 起動時あるいは再読み込み時には、画面が全て初期設定に戻ります。
- 上部にある
TogoDXのロゴをクリックすることでも、初期設定に戻ります。
Condition builderのClearボタンをクリックすると検索条件が初期化されます。



